朱方捷
发布时间:
2021-06-07
信息员:
|

教授 菌草生态产业协同创新中心副主任、福建省“闽江学者”特聘教授 Fish & Shellfish Immunology,CrystEngComm杂志审稿人 研究方向为开发、应用基于高通量测序与信息学分析的新技术,研究转录调控机制。
联系方式:
十大网投官方入口海峡联合研究院 350002
Email: fjzhu@fafu.edu.cn
实验室介绍: 转录系统生物学实验室:http://hbmcsysbio.team or /index.html 欢迎对统计和机器学习有较好理解的同学、同事加入学习、工作
学习及工作经历:
2003–2010 清华大学 学士,硕士
2010–2013 东京大学 博士 2014–2018 卡罗林斯卡医学院 博士后 2018–2020 剑桥大学 Research Associate 2020– 十大网投官方入口海峡联合研究院 教授
研究方向:
1)基因转录的调控机制
2)细胞命运决定与植物再生
3)基于高通量测序的技术开发
研究内容:
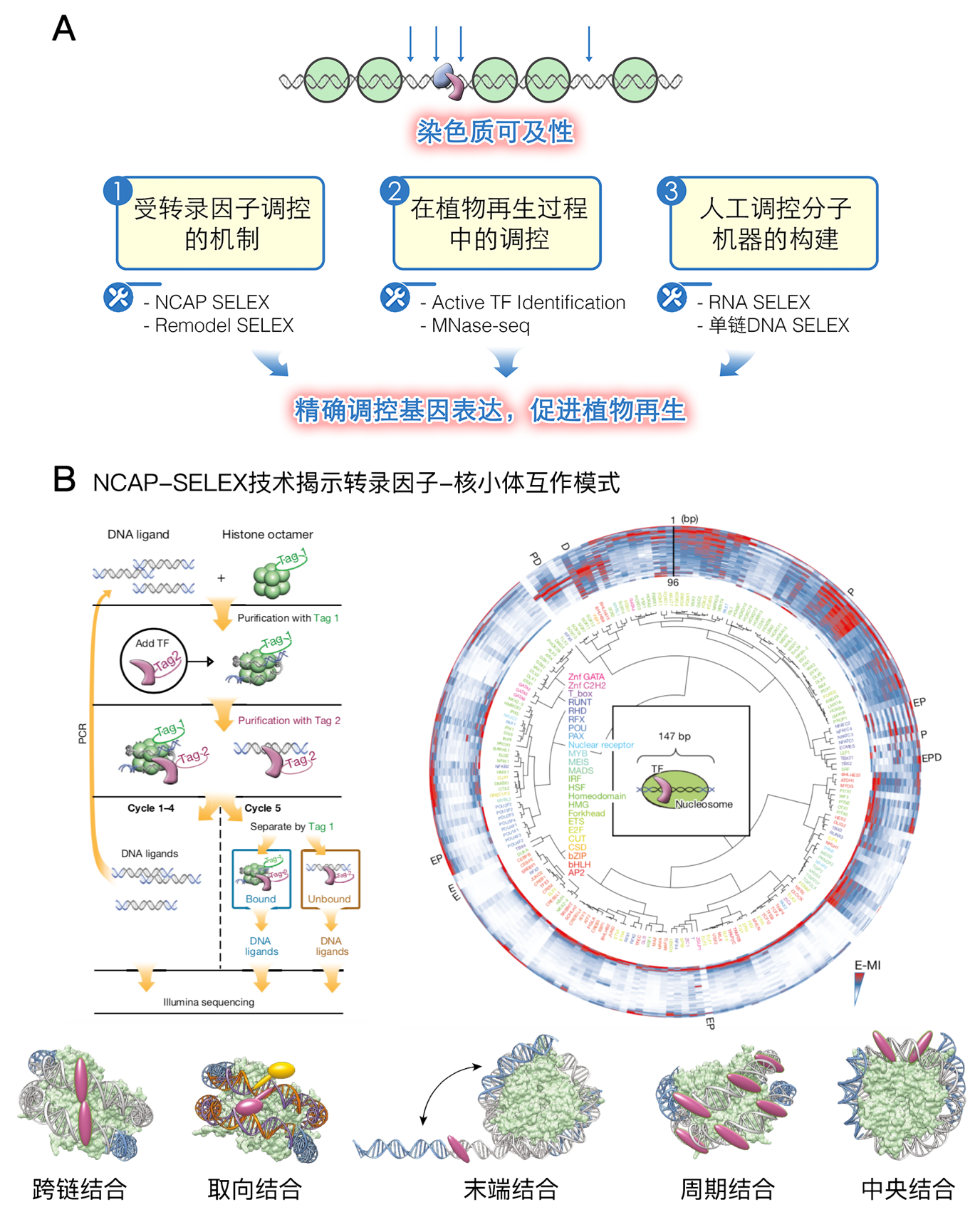
植物个体的所有细胞基因组相同,然而基因的差异表达导致了细胞命运的不同。我们结合超分子化学、表观基因组学、生物信息学等多学科研究手段,运用并开发基于测序和大数据的新型高通量生物技术,探讨植物基因表达的转录调控原理,解析植物细胞命运的决定机制。用于有效诱导植物再生,并贡献于植物基因编辑技术。 植物基因编辑技术在基础研究中是验证基因组位点功能的必备工具,在农业应用研究中广泛应用于作物的精准遗传改良。目前常用的CRISPR-Cas9技术利用农杆菌将Cas9核酸酶和gRNA导入到外植体中,编辑目的基因后,再通过添加生长素、激动素等因子将被编辑的植物组织诱导分化成完整植株。然而一些植物如香蕉、草莓、玉米中,组织再生的成功率低,导致基因编辑效率低下,阻碍了相关物种的深入研究。因此,阐明植物再生机制,探索提高组织再生效率的新途径,对植物学的理论和应用研究具有重要意义。 植物组织再生形成完整植株可通过器官从头再生或体细胞胚再生,再生过程包含脱分化与再分化等细胞命运的显著转变,分子水平体现为基因表达谱的全局性变化。干细胞研究表明,少数关键转录因子足以决定基因表达和细胞命运。在植物再生过程中,也有报导WUS、PLT、LEC、BBM等转录因子可调控细胞命运,诱导体细胞胚再生。然而,基因表达在真核生物中由转录因子与核小体两类主要元件协同控制,二者竞争性结合转录调控区域,决定染色质可及性与基因表达水平。植物再生时,转录因子结合、重塑、转移、修饰核小体的机制仍有待研究。因此,实验室的工作将围绕染色质可及性展开(图A)。我们计划应用各种表观基因组学技术如NCAP-SELEX(图B),Active TF identification, MNase-seq,ChIP-seq等,同时进行新技术研发,系统研究转录因子与核小体互作对转录过程和细胞命运的调控。
发表文章: F. Zhu, L. Farnung, E. Kaasinen, B. Sahu, Y. Yin, Bei. Wei, S.O. Dodonova, K.R. Nitta, E. Morgunova, M. Taipale, P. Cramer, J. Taipale, The interaction landscape between transcription factors and the nucleosome. Nature (IF = 41.577), 2018, 562, p76. F. Zhu, T. Nishimura and T. Kato, Organic/inorganic fusion materials: cyclodextrin-based polymer/CaCO3 hybrids incorporating dye molecules through host-guest interactions.Polym. J. (IF = 1.653), 2015, 47, p122. F. Zhu, T. Nishimura, H. Eimura, and T. Kato, Supramolecular effects on formation of CaCO3 thin films on a polymer matrix.CrystEngComm(IF = 3.304), 2014, 16, p1496. F. Zhu, T. Nishimura, T. Sakamoto, H. Tomono, H. Nada, Y. Okumura, H. Kikuchi, and T. Kato, Tuning the stability of CaCO3 crystals with magnesium ions for formation of aragonite thin films on organic polymer templates.Chem. Asian J.(IF = 3.692), 2013, 8, p3232. F. Zhu, Y. Kong, J. Liang, C. Li, Y. Hu, Y. Zhou, X. Liu, L. Xie, and R. Zhang, Tuning calcite magnesium content by soluble shell matrices: Insights into biomineral impurity control. Mater. Sci. Eng. C(IF = 5.080), 2010, 30, p963.
A. Jolma, J. Zhang, E. Mondragon, E. Morgunova, T. Kivioja, K.U. Laverty, Y. Yin,F. Zhu, G. Bourenkov, Q. Morris, T.R. Hughes, L.J.III Maher, J. Taipale, Binding specificities of human RNA-binding proteins toward structured and linear RNA sequences. Genome Res 2020, 30, p962. S.O. Dodonova, F. Zhu, C. Dienemann, J. Taipale, P. Cramer, Nucleosome-bound SOX2 and SOX11 structures elucidate pioneer factor function. Nature 2020, 580, p669. B. Wei, A. Jolma, B. Sahu, L.M. Orre, F. Zhong, F. Zhu, T. Kivioja, I. Sur, J. Lehtio, M. Taipale, J. Taipale, A protein activity assay to measure global transcription factor activity reveals determinants of chromatin accessibility. Nat. Biotechnol. 2018, 36, p521. E. Morgunova, Y. Yin, P.K. Das, A. Jolma, F. Zhu, A. Popov, Y. Xu, L. Nilsson, J. Taipale, Two distinct DNA sequences recognized by transcription factors represent enthalpy and entropy optima. Elife 2018, 7. J. Su, F. Zhu, G. Zhang, H. Wang, L. Xie and R. Zhang, Transformation of amorphous calcium carbonate nanoparticles into aragonite controlled by ACCBP. CrystEngComm, 2016, 18, p2125. T. Nishimura,F. Zhu, H. Tomono, H. Nada, T. Kato, バイオミネラリゼーションに学ぶ有機/無機複合体の設計と合成:高分子およびマグネシウムイオンによる炭酸カルシウムのモルホロジー制御, オレオサイエンス, 2014, 14, p417. H. Tomono, H. Nada, F. Zhu, T. Sakamoto, T. Nishimura, and T. Kato,Effects of magnesium ions and water molecules on the structure of amorphous calcium carbonate: a molecular dynamics study. J. Phys. Chem. B, 2013, 117,p14849. Y. Lin, W. Zhang, F. Zhu, J. Su, D. Fang, Y. Yang, G. Zhang, L. Xie, R. Zhang, and H. Wang, Subcellular localization of N-deoxyribosyltransferase in Lactobacillus fermentum: cell surface association of an intracellular nucleotide metabolic enzyme. FEMS Microbiol. Lett., 2011, 323, p132. L. Xie, F. Zhu, Y. Zhou, C. Yang, and R. Zhang, Molecular approaches to understand biomineralization of shell nacreous layer. Prog. Mol. Subcell. Biol., 2011, 52, p331. Y. Zhou, Z. He, C. Li, L. Xiang, F. Zhu, G. Zhang, L. Xie, and R. Zhang, Correlations among mRNA expression levels of Engrailed, BMP2 and Smad3 in mantle cells of pearl oyster Pinctada fucata. Prog. Biochem. Biophys., 2010, 37, p737. C. Li, J. Huang, S. Li, W. Fan, Y. Hu, Q. Wang, F. Zhu, L. Xie, and R. Zhang, Cloning, characterization and immunolocalization of two subunits of calcineurin from pearl oyster (Pinctada fucata). Comp. Biochem. Physiol. B: Biochem. Mol. Biol., 2009, 153, p43. Y. Kong, G. Jing, Z. Yan, C. Li, N. Gong, F. Zhu, D. Li, Y. Zhang, G. Zheng, H. Wang, L. Xie, and R. Zhang, Cloning and characterization of Prisilkin-39, a novel matrix protein serving a dual role in the prismatic layer formation from the oyster pinctada fucata. J. Biol. Chem., 2009, 284, p10841.
实验室成员: 温陈金(科研助理) 马娜娜(2020级博士研究生) 袁振(2020级硕士研究生) 李甜(2020级硕士研究生)
|
|

